Capitulo 6 VICAL en GEE
En esta sección se muestra cómo usar los archivos de VICAL para implementarlos en un script propio de GEE.
VICAL cuenta con tres archivos que puede importar a su Script, estos son:
// archivo para las colecciones de imagenes);
var imp = require('users/InifapCenidRaspa/VICAL:Exportaciones');
// archivo para los indices de vegetacion
var imp2= require('users/InifapCenidRaspa/VICAL:VegetationIndex');
// archivo para las visualizaciones
var St= require('users/InifapCenidRaspa/VICAL:Style');6.1 Coleccion de imagenes
Antes de importar el conjunto de colecciones de imágenes se deben definir ciertas variables que van a ser útiles para filtrar la colección, estos son: i) un punto o polígono; ii) intervalo de fechas y iii) valor umbral de nubes en las imágenes; esto se muestra a continuación:
var fecha = ['2021-01-01', '2022-03-18']; //Fecha inicial y final
//poligono o punto
var table = ee.FeatureCollection("projects/calcium-verbena-328905/assets/Bate");
var p_nubes= 30;//Porcentaje de nubes6.1.1 Landsat
Si se desea usar imágenes LandSat (4, 5, 7, 8 y 9) corregidas atmosféricamente libre de nubes se puede usar el código siguiente, donde se crea una función para unir las colecciones de imágenes. Para ello se usa el archivo imp.
function ColeccionImagenSR(fecha, recorte, umbral)
{
// se importan las colecciones de imagenes usando el archivo "imp"
var L9sr = imp.ColeccionLandsatSR(fecha, 'LC09', recorte, umbral);
var L8sr = imp.ColeccionLandsatSR(fecha, 'LC08', recorte, umbral);
var L7sr = imp.ColeccionLandsatSR(fecha, 'LE07', recorte, umbral);
var L5sr = imp.ColeccionLandsatSR(fecha, 'LT05', recorte, umbral);
var L4sr = imp.ColeccionLandsatSR(fecha, 'LT04', recorte, umbral);
//Los datos de ETM y ETM+ se ajustan espectrales a OLI y OLI-2
var L7a = L7sr.map(imp.TMaOLI);
var L5a = L5sr.map(imp.TMaOLI);
var L4a = L4sr.map(imp.TMaOLI);
// Une las tres series de imágenes
var serieT =L9sr.merge(L8sr).merge(L7a).merge(L5a).merge(L4a).sort('system:time_start');
return serieT;
}
//SE importa la coleccion usando la funccion anterior
var l8Sergio=ColeccionImagenSR(fecha, table, p_nubes);
//podemos imprimir las imagenes usando la funcion print() para ver
//si se llevo a cabo el filtrado de la coleccion de imagenes (Figura 6.1)
print (l8Sergio);Con estas colecciones de imágenes se pueden calcular series de tiempo de diferentes índices de vegetación.
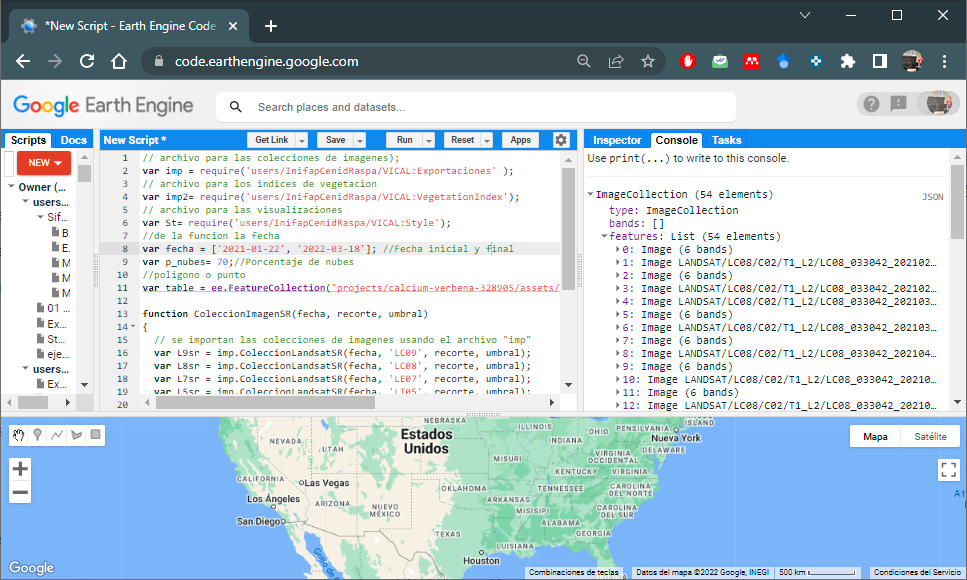
Figura 6.1: Impresion de coleccion de imagenes Landsat
6.1.2 Sentinel-2
Si se desea usar imágenes Sentinel-2 corregidas atmosféricamente libre de nubes se puede usar el siguiente código.
//SE importa la coleccion usando el siguiente codigo
var S2sr = imp.ColeccionImagenSentinelSR(fecha, table, p_nubes);
//podemos imprimir las imagenes usando la funcion print() para ver
//si se llevo a cabo el filtrado de la coleccion de imagenes (Figura 6.2)
print (S2sr);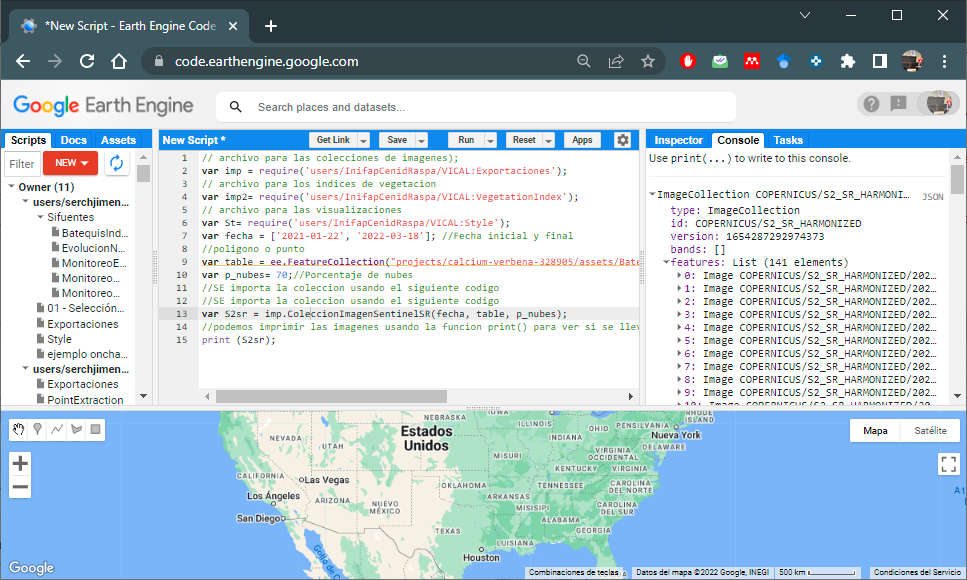
Figura 6.2: Impresion de coleccion de imagenes Sentinel-2
6.1.3 Landsat y Sentinel-2
Si se desea usar imágenes LandSat y Sentinel-2 corregidas atmosféricamente libre de nubes se puede usar el siguiente código, los datos se ajustaron espectralmente a las bandas espectrales de Landsat 8. Se emplean las funciones descritas en la Sección 6.1:
function ColeccionImagenAMBOS(fecha, recorte, umbral)
{
//Se lee la function para imagenes Landsat con ajuste espectral
var L8Conjunto=ColeccionImagenSR(fecha, recorte, umbral)
//coleccion sentinel
var S2sr = imp.ColeccionImagenSentinelSR(fecha, recorte, umbral);
var S2a = S2sr.map(imp.MSIaOLI); //Se hace el ajuste espectral de sentinel-2 a Landsat
var serieT = S2a.merge(L8Conjunto).sort('system:time_start');
return serieT;
}
//SE importa la coleccion usando el siguiente codigo
var S2B = ColeccionImagenAMBOS(fecha, table, p_nubes);
//podemos imprimir las imagenes usando la funcion print() para ver
//si se llevo a cabo el filtrado de la coleccion de imagenes (Figura 6.3)
print (S2sr);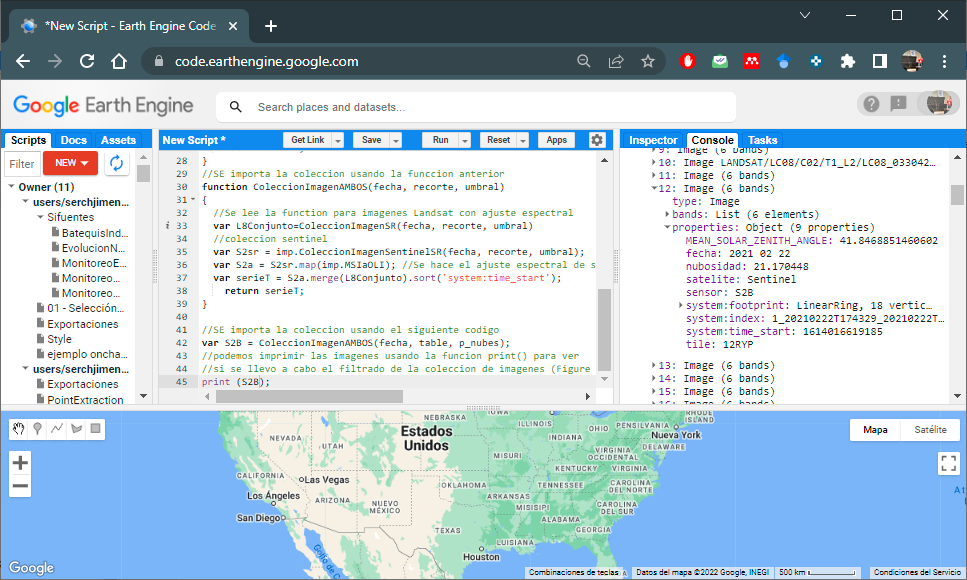
Figura 6.3: Impresion de coleccion de imagenes Landsat y Sentinel-2
Para revisar un Script de ejemplo clic aquí
6.2 Indices de vegetación
Para usar el código de índices de vegetación de VICAL se tienen que usar el archivo imp2; y se mandan a llamar estos IV usando los nombres de la columna ExpresionGEE que se muestran en la Tabla 6.1.
Por ejemplo, para calcular el NDVI con la colección de imágenes LandSat y Sentinel-2 de la sección 6.1.3 se usaría el siguiente código:
//Normalized Difference Vegetation Index- NDVI
var ivs = ee.ImageCollection(S2B.map(imp2.NDVI));
//para imprimir y ver la banda de NDVI
print (ivs);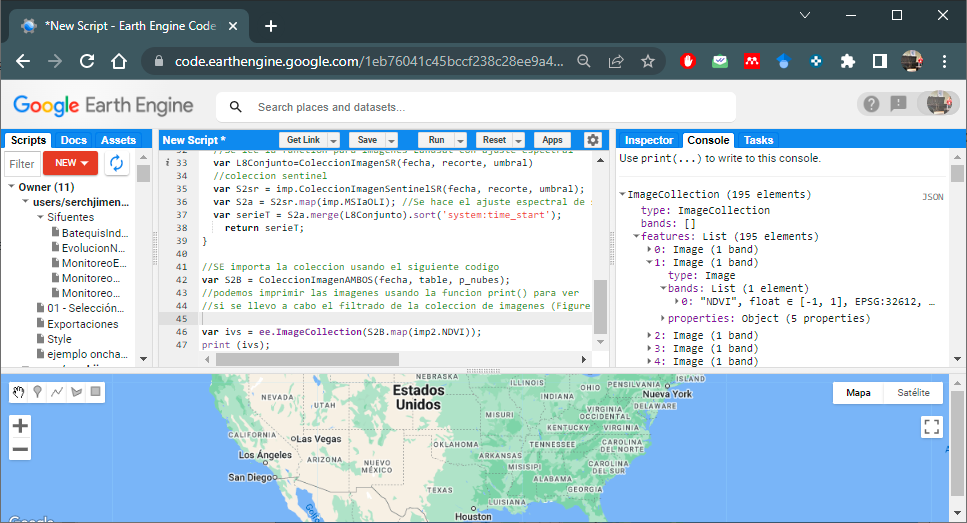
Figura 6.4: Impresion de coleccion de imagenes con banda NDVI
Ahora, para visualizar en el mapa el NDVI de la primera imagen de la colección y recortado para la zona quedaría como se muestra en el código siguiente; donde para ver la paleta de colores se utilizaría el archivo st de ¨VICAL.
//Se obtiene el NDVI de la primera imagen en la coleccion
var iv = ivs.first();
//Paleta de colores donde se usa el archivo st
var ivVis = {min :0, max : 1, palette : St.paletaIV};
Map.addLayer(iv.clip(table), ivVis,'NDVI'); //Indice
//se centra el mapa a la zona
Map.centerObject(table, 13);En la Figura 6.5. se muestra el mapa de NDVI para la zona de interés.
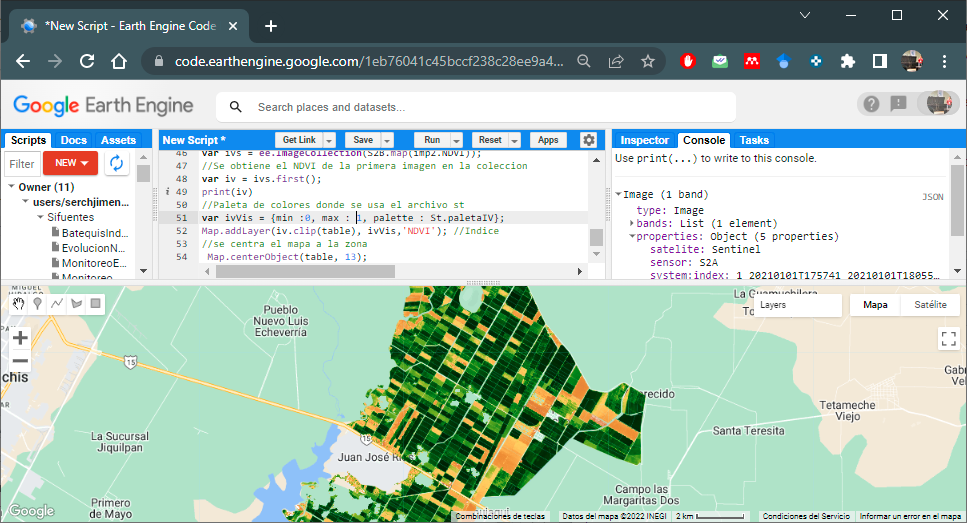
Figura 6.5: mapa de NDVI para el area de interes
Para revisar el codigo del ejemplo clic aquí
Si se desea visualizar el NDVI de una imagen en particular se debe convertir a lista de después mandar a llamarlo.
| Numero | Indice | Abreviatura | ExpresionGEE | Coeficientes |
|---|---|---|---|---|
| 1 | Atmospherically resistant vegetation index | ARVI* | ARVI | γ=1.0 |
| 2 | Adjusted transformed soil-adjusted vegetation index | ATSAVI* | ATSAVI | |
| 3 | Difference vegetation index | DVI | DVI | |
| 4 | Enhanced vegetation index | EVI | EVI | C1=6.0, C2= 7.5; L=1.0 |
| 5 | Enhanced vegetation index | EVI2* | EVI2 | C1=2.4 |
| 6 | Green normalized difference vegetation index | GNDVI | GNDVI | |
| 7 | Modified soil adjusted vegetation index | MSAVI2 | MSAVI2 | |
| 8 | Moisture stress index | MSI | MSI | |
| 9 | Modified triangular vegetation index | MTVI | MTVI | |
| 10 | Modified triangular vegetation index-2 | MTVI2 | MTVI2 | |
| 11 | Normalized difference tillage index (NDTI) | NDTI | NDTI | |
| 12 | Normalized difference vegetation index | NDVI | NDVI | |
| 13 | Normalized difference water index | NDWI | NDWI | |
| 14 | Optimized soil adjusted vegetation index | OSAVI* | OSAVI | X=0.16 |
| 15 | Renormalized difference vegetation index | RDVI | RDVI | |
| 16 | Redness index | RI | RI | |
| 17 | Ratio vegetation index | RVI | RVI | |
| 18 | Soil adjusted vegetation index | SAVI* | SAVI | L=0.5 |
| 19 | Triangular vegetation index | TVI | TVI | |
| 20 | Transformed soil adjusted vegetation index | TSAVI* | TSAVI | a= 1 ; b=0; |
| 21 | Visible atmospherically resistant index | VARI | VARI | |
| 22 | Vegetation index number or simple ratio | VIN | VIN | |
| 23 | Wide dynamic range vegetation index | WDRVI* | WDRVI | α=0.2 |